PCR-testingen for Sars-CoV-2 som diagnostisk verktøy er et svært sentralt element hvis man skal forstå samt evaluere pandemiens forløp. En av de viktigste og grundigste gjennomgangene av test-protokollen med PCR for Sars-CoV-2 er Corman Drosten Review. WHOs testprotokoll (og også Norges) for Sars-CoV-2 baserte seg på rapporten til Drosten et al. som ble publisert hos det medisinske tidsskriftet Eurosurveillance, og CormanDrostenReview er evalueringen og kritikken av denne rapporten.
Foreningen Lov og Helse har i samarbeid med flere fagpersoner oversatt den til norsk. Originalen på engelsk kan du lese blant annet på cormandrostenreview.com.
Nedenfor følger vår oversettelse:
Evalueringsrapport av RT-PCR-protokollen for Sars-CoV-2 av Corman-Drosten et al.
27. november 2020.
Denne omfattende evalueringsrapporten ble 27. november offisielt oversendt redaksjons-komiteen ved Eurosurveillance, via deres innsendingsportal. Vedlagt denne evalueringsrapporten er et brev med anmodning om tilbaketrekning, undertegnet av samtlige hoved- og medforfattere. Det første og siste navnet på forfatterlisten er den første og andre hovedforfatteren. Alle navn mellom disse er medforfattere.
Ekstern fagfellevurdering av RT-PCR testen for å oppdage SARS-CoV-2 avslører 10 alvorlige feil på molekylært og metodologisk nivå: med falske positive resultater som konsekvens.
Pieter Borger(1), Bobby Rajesh Malhotra(2), Michael Yeadon(3), Clare Craig(4), Kevin McKernan(5), Klaus Steger(6), Paul McSheehy(7), Lidiya Angelova(8), Fabio Franchi(9), Thomas Binder(10), Henrik Ullrich(11), Makoto Ohashi(12), Stefano Scoglio(13), Marjolein Doesburg-van Kleffens(14), Dorothea Gilbert(15), Rainer Klement(16), Ruth Schruefer(17), Berber W. Pieksma(18), Jan Bonte(19), Bruno H. Dalle Carbonare(20), Kevin P. Corbett(21), Ulrike Kämmerer(22)
SAMMENDRAG
I publikasjonen med tittel “Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR” (Eurosurveillance 25(8) 2020)[1] presenterer forfatterne en diagnostisk prosedyre og RT-q PCR-protokoll for oppdagelse og diagnostisering av 2019-nCoV (nå kjent som SARS-CoV-2), som de hevder har blitt validert samt å være en robust diagnostisk metodologi for laboratoriebruk i offentlig helsevesen.
Med tanke på alle konsekvensene denne publikasjonen har medført for samfunn over hele verden, har en gruppe uavhengige forskere gjort en punktvis gjennomgang av publikasjonen der 1) alle komponenter i det presenterte test-designet ble kryss-sjekket, 2) RTq-PCR protokoll-anbefalingene ble vurdert i forhold til good laboratory practice, og 3) parameterne ble undersøkt mot relevant vitenskapelig litteratur på fagområdet.
Den publiserte RTq-PCR-protokollen for oppdagelse og diagnostisering av 2019-nCoV, samt manuskriptet, lider under flere tekniske og vitenskapelige feil, inklusive utilstrekkelig primer-design, en problematisk og utilstrekkelig RTq-PCR-protokoll, og fraværet av en tilstrekkelig test-validering. Verken den beskrevne testprotokollen eller manuskriptet i seg selv oppfyller kravene til en akseptabel vitenskapelig publikasjon. Videre er alvorlige interessekonflikter hos forfatterne ikke omtalt. Og til sist viser den svært korte tidsperioden mellom innsending og godkjenning av publikasjonen (24 timer) at en systematisk fagfelle-vurderingsprosess enten ikke ble gjennomført her, eller var av bekymringsverdig lav kvalitet. Vi fremlegger udiskutable bevis på flere vitenskapelige feil og mangler.
Tatt i betraktning de vitenskapelige og metodologiske feiltrinn som presenteres her, føler vi oss trygge på at redaksjonsrådet til Eurosurveillance ikke har annet valg enn å trekke tilbake denne publikasjonen.
KORT GJENNOMGANG AV DOKUMENTET
Dette dokumentet vil vise til flere alvorlige feil i Corman-Drosten-rapporten, og at resultatet av disse har ført til verdensomspennende feildiagnoser av infeksjoner som tilskrives SARS-CoV-2 og som blir knyttet til sykdommen COVID-19. Vi er blitt konfrontert med strenge nedstengninger som har ødelagt liv og livsgrunnlag for mange mennesker, begrenset adgangen til utdannelse, og disse regjerings-pålagte restriksjonene rundt om i verden er et direkte angrep på folkets grunnleggende rettigheter og deres personlige friheter, og har medført følgeskader for nasjonale økonomier på global skala.
Det er ti store problemer med Corman-Drosten-rapporten som vi vil beskrive og forklare mer detaljert i de følgende avsnittene.
Det første hovedtemaet er at det nye koronaviruset SARS-CoV-2 (som i publikasjonen er kalt 2019-nCoV og som i februar 2020 ble kalt SARS-CoV-2 av en internasjonal gruppe virus-eksperter) er basert på in silico (teoretiske) sekvenser, skaffet til veie av et laboratorium i Kina [1], fordi på dette tidspunktet var verken kontrollmateriale av smittsomt («live») Sars-CoV-2 eller fragmenter derav tilgjengelig. Det eksisterte heller ikke noe isolert genomisk RNA av viruset som var tilgjengelig for forfatterne. Til dags dato har ingen validering blitt utført av forfatterne basert på isolerte SARS-CoV-2 virus eller full-lengde RNA av disse. Ifølge Corman et al.:
“Vår målsetting var å utvikle og distribuere robust diagnostisk metodologi til bruk for laboratorie-miljøer i offentlig helsetjeneste, uten å ha tilgang til virus-materiale.» [1]
Her bør fokus være på de to uttrykte målsetningene: a) utvikling og b) distribuering av en diagnostisk test til bruk for laboratorie-miljøer i offentlig helsetjeneste. Disse målsetningene kan ikke oppnås uten tilgang til det aktuelle virus-materialet (f. eks. for å bestemme smittsom virusmengde). Bare en protokoll med maksimal nøyaktighet kan være den obligatoriske og primære målsetningen. Å bestemme kritisk virusmengde er helt nødvendig informasjon, og det er Christian Drosten’s gruppe som er ansvarlig for å utføre disse eksperimentene og skaffe til veie denne type avgjørende og viktige data.
Til tross for manglene ble altså disse in silico sekvensene brukt til å utvikle en RT-PCR test-metodikk for å identifisere det nevnte viruset. Denne modellen var basert på antakelsen om at det nye viruset er svært likt SARS-CoV fra 2003, ettersom begge er beta-koronavirus.
PCR testen ble derfor designet ved bruk av genomsekvensen til SARS-CoV som kontrollmateriale for Sarbeco-komponenten; vi kjenner til dette ut fra vår personlige epost-kommunikasjon med en av medforfatterne til Corman-Drosten-rapporten [2]. Metoden for å modellere SARS-CoV-2 ble beskrevet i Corman-Drosten-rapporten som:
«Etablering og validering av en diagnostisk workflow for 2019-nCoV screening og spesifikk påvisning, designet i fraværet av tilgjengelige virus-isolater eller originale pasient-prøver. Design og validering var mulig gitt den nære genetiske beslektelse til 2003 SARS-CoV, og ved hjelp av syntetisk nukleinsyre-teknologi.»
Reversert transkripsjon-polymerase kjedereaksjon (RT-PCR) er en viktig biomolekylær teknologi for hurtig identifisering av små RNA-fragmenter, som er kjent på forhånd. I første trinn blir enkeltrådet RNA-molekyler som finnes i prøven revers-transkribert (kopiert) for å danne dobbeltrådet cDNA. Dette cDNA blir deretter amplifisert ved bruk av et spesifikt primer-par og et termostabilt DNA-polymerase-ensym. Denne teknologien er svært følsom og dens deteksjonsgrense er i teorien begrenset til så lite som bare 1 molekyl av cDNA. Spesifisiteten av PCR er imidlertid sterkt påvirket av biomolekylære designfeil.
Hva er viktig når man designer en RT-PCR test, og en kvantitativ RT-qPCR test slik som beskrevet i Corman-Drosten-rapporten?
- Primere og prober:
a) Konsentrasjonen av primerne og prober må være innenfor et optimalt spenn (100-200 nM)
b) Må være spesifikke for et målgen som ønskes amplifisert
c) Må ha en optimal prosentandel av GC-innhold i forhold til totalen av nitrogenbaser (minimum 40%, maksimum 60%)
d) For virus-diagnostisering må minst tre primerpar kunne detektere tre virale gener (helst lengst mulig fra hverandre i det virale genomet)
2. Temperaturen der alle de biokjemiske prosesser foregår:
a) DNA-smeltetemperatur (>92°)
b) DNA-amplifikasjonstemperatur (spesifikk for TaqPol)
c) Tm, smeltetemperaturen (temperaturen der primere og prober hybridiserer til/separeres fra mål-DNA; skal ikke overstige 2 ̊C per primerpar). Tm er sterkt avhengig av GC-innholdet i primerne
3. Antall amplifikasjons-sykler (færre enn 35, helst 25-30 sykler)
Ved viruspåvisning vil mer enn 35 sykler oppdage signaler som ikke er i samsvar med smittsomt virus, hvilket som blir bestemt ved isolering i cellekultur (gjennomgått i [2]); dersom noen tester PCR-positivt der en terskelverdi på 35 sykluser eller mer er benyttet (som er tilfellet i de fleste laboratorier i Europa og USA), er sannsynligheten for at denne personen faktisk er smittet mindre enn 3%, mens sannsynligheten for at nevnte resultat er en falsk positiv er 97% (gjennomgått i [3]).
4. Molekylære biologiske valideringer; amplifiserte PCR-produkter må bli validert enten ved å kjøre produktene i en gel med en DNA-linjal, eller ved direkte DNA-sekvensering.
5. Positive og negative kontroller bør spesifiseres for å bekrefte/avkrefte spesifikk virus-deteksjon
6. En standard operasjonsprosedyre (SOP) bør være tilgjengelig
En SOP skal spesifisere tydelig de ovenstående parametere, slik at alle laboratorier er i stand til å sette opp nøyaktig like testforhold. Det er helt vesentlig å ha en validert universell SOP, fordi det muliggjør sammenlikning av data, internt og mellom forskjellige land.
ANDRE MINDRE PROBLEMER MED CORMAN-DROSTEN-RAPPORTEN
1. I Tabell 1 i Corman-Drosten-rapporten er forskjellige forkortninger benyttet -«nM» er spesifisert, men ikke «nm». Videre, med hensyn til korrekt nomenklatur, så betyr nm «nanometer», derfor burde nm vises som nM her.
2. Det er generell konsensus alltid å skrive genetiske sekvenser i 5’-3’ retningen, inklusive revers-primerne. Det er svært uvanlig å gjøre en justering med reversert komplementær skriving av primersekvensen, slik forfatterne gjorde i figur 2 i Corman-Drosten-rapporten. Her er i tillegg en uspesifikk base («wobble base») markert som «y» uten beskrivelse av hvilke baser som Y betegner.
3. To villedende fallgruver i Corman-Drosten-rapporten er at deres Tabell 1 ikke inkluderer Tm-verdier (annealingtemperaturer). Den viser heller ikke GC-verdier (antall G og C i sekvensene som %-verdi av totale baser).
ALVORLIGE PROBLEMER MED CORMAN-DROSTEN-RAPPORTEN
A) BAKGRUNN
Forfatterne introduserer bakgrunnen for deres vitenskapelige arbeid slik: «Det pågående utbruddet av det nye coronaviruset (2019-nCoV) som nylig er dukket opp, utgjør en utfordring for laboratorier i offentlig helsevesen ettersom virus-isolater ikke er tilgjengelig, samtidig som det er økende dokumentasjon på at viruset er mer utbredt enn opprinnelig antatt, og at internasjonal spredning via reisende allerede forekommer.»
I følge BBC News [4] og Google Statistics [5] var det 6 dødsfall globalt den 21. januar 2020 – dagen da manuskriptet ble innsendt. Hvorfor antok forfatterne at det ville bli en utfordring for laboratorier i offentlig helsevesen når det ikke fantes dokumentasjon av betydning på det tidspunktet som indikerte at utbruddet var mer omfattende enn opprinnelig antatt?
Forfatterne har uttrykt en målsetting om å utvikle og implementere en robust diagnostisk metodologi for bruk i laboratorie-miljøer i offentlig helsevesen, uten tilgjengelig virusmateriale. De erkjenner videre at «Den foreliggende studien viser den enorme responskapasiteten som er oppnådd gjennom koordinering av akademiske og offentlige laboratorier i europeiske forskningsnettverk.»
B) METODER OG RESULTATER
1. Primer og probe-design
1a) Feilaktige primer-konsentrasjoner
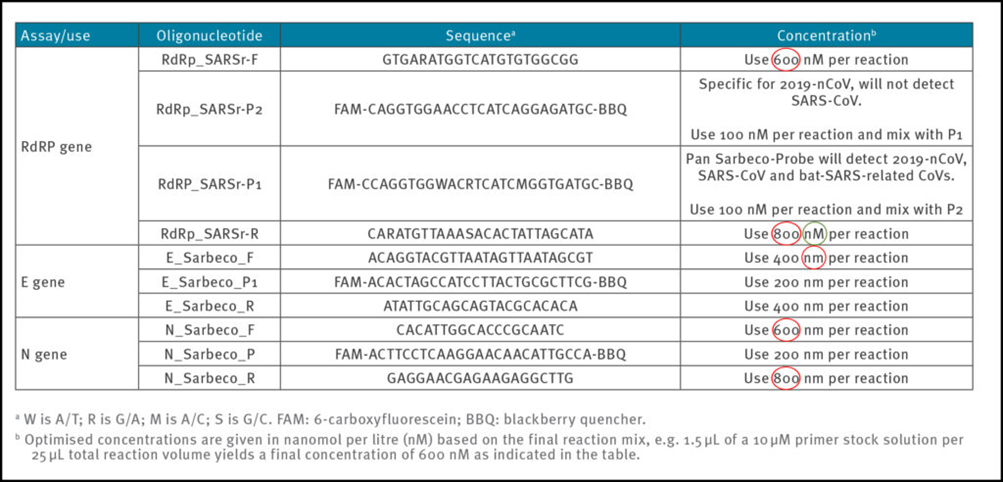
Pålitelige og nøyaktige PCR-test protokoller blir vanligvis designet med bruk av mellom 100nM og 200nM per primer (7). I Corman-Drosten-rapporten kan vi se uvanlig høye og varierende primer-konsentrasjoner for flere primere (tabell 1). For RdRp_SARSr-F og RdRp_SARSr-R primerparene er henholdsvis 600 nM og 800 nM beskrevet. Likeledes, for N_Sarbeco_F og N_Sarbeco_R primer-settet anbefaler de henholdsvis 600 nM og 800 nM [1].
Det burde være åpbenbart at disse konsentrasjonene er altfor høye til å være optimale for spesifikke kopieringer av målgener. Det finnes ingen oppgitt grunn for å bruke disse ekstremt høye konsentrasjonene av primere i protokollen. Det er heller slik at disse konsentrasjonene fører til økt uspesifisert binding og amplifikasjon av PCR-produktet.
Tabell 1: Primere og prober (etter Corman-Drosten-rapporten; feilaktige primer-konsentrasjoner er uthevet)
1b) Uspesifikke (wobbly) primer- og probe-sekvenser
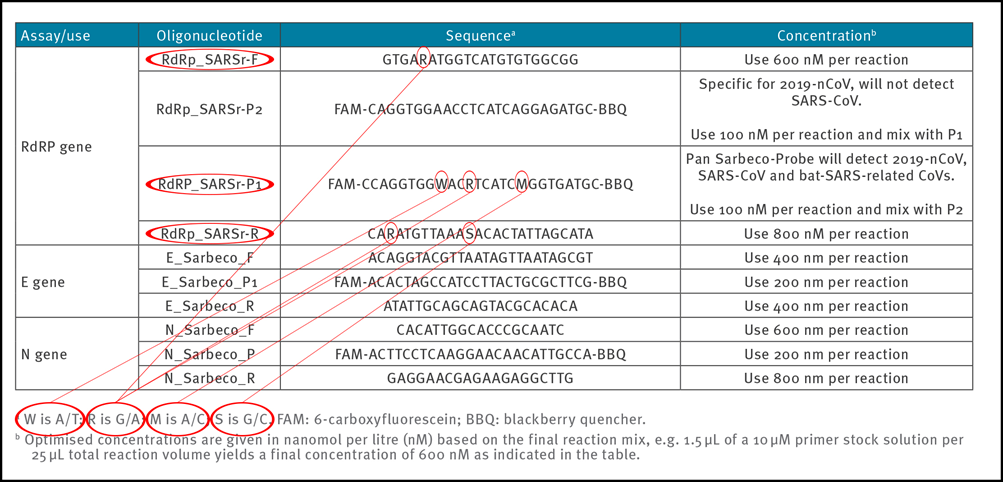
For å oppnå reproduserbare og sammenliknbare resultater er det vesentlig å definere primer-parene klart. I Corman-Drosten-rapporten ser vi seks uspesifiserte posisjoner, indikert ved bokstavene R, W, M og S (tabell 2). Bokstaven W betyr at denne posisjonen kan enten være en A eller en T; R betyr at posisjonen kan være en G eller en A, M betyr at posisjonen kan være enten en A eller en C, bokstaven S kan være en G eller en C i denne posisjonen. Dette høye antallet av varianter er ikke bare uvanlig, men også svært forvirrende for laboratorier. Disse seks uspesifiserte posisjonene kan lett resultere i at det blir designet flere forskjellige alternative primersekvenser som ikke relaterer til SARS-CoV-2 (2 ulike RdRp_SARSr_F primere + 8 ulike RdRp_SARS_P1 prober + 4 ulike RdRp_SARSr_R). Variasjonene i design vil uunngåelig føre til resultater som ikke engang er SARS- CoV-2-relatert. Den forvirrende uspesifiserte beskrivelsen i Corman-Drosten-rapporten er derfor ikke egnet som standard operasjons-prosedyre (SOP). Disse uspesifiserte posisjonene burde ha vært utvetydig bestemt.
Disse uspesifikke sekvensene er allerede blitt en kilde til bekymring i fagmiljøet, og har resultert i et «Letter to the Editor» skrevet av Pillonel et al. [8], angående helt åpenbare feil i de beskrevne sekvensene. Disse feilene vises også i tilleggsmaterialet som følger med rapporten hos Corman et al.
Tabell 2: Primere og prober (etter Corman-Drosten-rapporten; uspesifiserte (wobbly) nukleotider i primerne er uthevet)
WHO-protokollen (figur 1), som stammer direkte fra Corman-Drosten-rapporten, konkluderer med at for å bekrefte forekomst av SARS-CoV-2 må to kontrollgener (E-genet og RdRp-genet) være identifisert i analysen. Man bør merke seg at RdRp-genet har en usikker posisjon («wobbly») i forward-primeren (R=G/A), to usikre posisjoner i reversprimeren (R=G/A; S=G/C) og tre usikre posisjoner i RdRp-proben (W=A/T; R=G/A; M=A/C). Altså, to forskjellige forward-primere, fire forskjellige reversprimere, og at åtte ulike prober kan syntetiseres for RdRp-genet. Alt i alt er det 64 mulige kombinasjoner av primerne og prober!
Corman-Drosten-rapporten identifiserer i tillegg et tredje gen som, ifølge WHO-protokollen, ikke ble ytterligere validert og ansett som unødvendig:
«Legg merke til at N-gen-analysen også presterte bra, men ble ikke gjenstand for ytterligere intensiv validering, fordi den var litt mindre følsom.»
Dette var en uheldig utelatelse fordi det ville være best å benytte alle tre gen-PCRene som bekreftende analyser, og dette ville ha resultert i et nesten tilstrekkelig diagnostisk testprotokoll for virus RNA-deteksjon. Tre bekreftende analyse-trinn ville i det minste minimere vekk feil og unøyaktigheter ved hvert trinn med hensyn til de uspesifiserte posisjonene («wobbly spots»). (Ikke desto mindre ville protokollen likevel komme til kort i forhold til enhver såkalt GLP «good laboratory practice», når man inkluderer alle de andre designfeilene).
Slik det står, så er beklageligvis N-gen-analysen verken foreslått i WHO-anbefalingen (figur 1) som et obligatorisk og essensielt tredje bekreftende trinn, eller lagt vekt på i Corman-Drosten-rapporten som en mulig og viktig kryss-validering for en «rutinemessig workflow» (tabell 2).
Som følge av dette ble bare to primersett benyttet i stedet for alle tre, i nesten alle test-prosedyrer over hele verden. Denne forglemmelsen gjør hele testprotokollen ubrukelig med hensyn til å gi nøyaktige testresultater av reell betydning i en pågående pandemi.
Figur 1: Analysen for å bekrefte N-genet er verken lagt vekt på som et nødvendig tredje trinn i den offisielle WHO Drosten-Corman protokoll-anbefalingen under [8], og er heller ikke påkrevet som et avgjørende trinn for bedre testnøyaktighet i Eurosurveillance-publikasjonen.

1c) Feilaktig GC-innhold (omtalt i 2c, sammen med smeltetemperatur (Tm))
1d) Deteksjon av virale gener
RT-PCR er ikke anbefalt for primær-diagnose av infeksjon. Dette er grunnen til at RT-PCR-testen som benyttes i klinisk rutine for deteksjon av COVID-19 ikke er indikativ for en COVID-19 diagnose på regulatorisk grunnlag.
«Klinikere trenger å være klar over den forbedrede nøyaktighet og hastighet i de molekylære diagnostiske teknikkene ved diagnostisering av infeksjoner, men også deres begrensninger. Laboratorie-resultater bør alltid bli tolket i kontekst av den kliniske presentasjonen av pasienten, og korrekt sted, kvalitet og timing når prøver bliver tatt er nødvendig for pålitelige resultater.» [9]
Imidlertid kan PCR-testen være verdifult for legene til å differensiere mellom diagnosene når han eller hun må skille mellom forskjellige lungeinfeksjoner (influensa, Covid-19 og SARS har svært like symptomer). For en bekreftende diagnose av et spesifikt virus må minst tre spesifikke primerpar benyttes for å finne tre virus-spesifikke gener. Disse målgenene bør helst være lokalisert med størst mulig avstand i det virale genomet (inkludert motsatte ender).
Selv om Corman-Drosten-rapporten beskriver tre primere, så dekker disse primerne bare omtrent halvparten av virusets genom. Dette er enda en faktor som reduserer spesifisiteten for deteksjon av intakt COVID-19 virus-RNA, og øker andelen av falske positive testresultater.
Derfor, selv om vi oppnår tre positive signaler (det vi si tre primerpar gir tre forskjellige amplifikasjonsprodukter) i en prøve, så beviser ikke dette forekomst av et virus. Et bedre primer-design ville bestå av ende-primerne, i begge ender av det virale genomet. Dette fordi hele det virale genomet da ville vært dekket, og tre positive signaler kan skille bedre mellom et komplett (og dermed potensielt smittsomt) virus og fragmenterte virale genomer (uten smittepotensial). For å trekke noen betydningsfull konklusjon om smittsomheten av viruset, bør Orf1-genet, som koder for det essensielle replikerende enzymet (replicase enzyme) til SARS-CoV-viruset, være inkludert som et målgen (figur 2). Plasseringen av målene i området i det virale genomet som er oftest og mest variabelt transkribert er en annen svakhet ved protokollen.
Kim et al. påviser meget variabel 3’ ekspressjon av subgenomisk RNA i Sars-CoV-2 [23]. Disse RNA-fragmentene, som spesifikke indikatorer, er aktivt overvåket i asymptomatiske og ikke-smittebærende pasienter [10]. Det er meget betenkelig å screene en befolkning av asymptomatiske personer med qPCR primere som har en 6-basepar primer-dimer i 3’-primer enden av en primer (figur 3). Tilsynelatende anbefaler WHO disse primerne. Vi testet alle «wobble»-derivatene fra Corman-Drosten-rapporten med Thermofisher’s primer-dimer online-verktøy [11]. RdRp forward-primeren har en 6bp 3’-prime-homologi med Sarbeco E Reverse-primeren. Ved en høy primer-konsentrasjon kan dette gi unøyaktigheter.
I tillegg: Det er perfekt overensstemmelse med én av N-gen-primerne og et klinisk patogen (Pantoea), som finnes hos immunsvekkede pasienter. Revers-primeren treffer også Pantoea, men ikke i det samme målområdet (figur 3).
Dette er alvorlige designfeil, ettersom testen ikke kan skille mellom hele viruset og virale fragmenter. Testen kan derfor ikke brukes for diagnostisering av SARS-virus.
Figur 2: Relative posisjoner for målgener på SARS koronaviruset og det nye 2019 koronavirusgenomet. ORF: open reading frame; RdRp: RNA-dependent RNA polymerase. Tall under målgenene er genomposisjoner i henhold til SARS-CoV, NC_004718 [1]

Figur 3: En test med Thermofischer’s primer-dimer online-verktøy viser at RdRp forward-primeren har 6bp i 3‘prime-enden som samsvarer med Sarbeco E Reverse-primeren (venstre boks). En annen test viser at det er perfekt overensstemmelse mellom én av N-gen-primere og et klinisk patogen (Pantoea), som finnes hos immunsvekkede pasienter (høyre boks).

2. Reaksjonstemperaturer
2a) DNA-smeltetemperatur (>92°).
Tilfredsstillende behandlet i Corman-Drosten-rapporten.
2b) DNA-amplifikasjonstemperatur.
Tilfredsstillende behandlet i Corman-Drosten-rapporten.
2c) Feilaktig GC-innhold og Tm
Annealings-temperaturen bestemmer ved hvilken temperatur primerne hybridiserer til/separeres fra målsekvensen. For en effektiv og spesifikk amplifikasjon bør GC-innholdet i primerne nå et minimum av 40% og et maksimum av 60%. Som antydet i tabell 3, så er tre av primerne, som beskrives i Corman-Drosten-rapporten, ikke innenfor de optimale grensene for GC-innhold. To primere (RdRp_SARSr_F og RdRp_SARSr_R) har uvanlige og svært lave GC-verdier på 28 % – 31 % for alle mulige varianter av uspesifikke baser («wobble»-baser), mens primeren E_Sarbeco_F har en GC-verdi på 34.6 % (tabell 3 og andre panel i tabell 3).
Det er viktig å vite at GC-innholdet i stor grad bestemmer bindingen til primerens spesifikke mål, fordi disse baseparene hybridiserer med tre hydrogen-bindinger. Det vil si at jo mindre GC-innhold i primeren, desto lavere er dets evne til å binde seg til den spesifikke sekvensen i målgenet (altså til genet som skal oppdages). For å muliggjøre en hybridisering med en målsekvens, må det velges en temperatur som er så nær som mulig til den faktiske annealings-temperaturen («best practice value») for at primere ikke skal separere seg fra målet igjen, samtidig som hybridiseringen må være målrettet til den spesifikke sekvensen.
Dersom Tm-verdien er meget lav, slik det er tilfellet for alle de uspesifikke varianter av RdRp revers-primere, kan primere binde seg ikke-spesifikt til flere mål, noe som reduserer spesifisiteten og øker potensiale for falske positive resultater.
Annealings-temperaturen (Tm) er en avgjørende faktor for å fastslå spesifisiteten/nøyaktigheten av qPCR-prosedyren, og essensiell for å bedømme nøyaktigheten av qPCR-protokoller. Best-practice anbefaling er: begge primere (forward– og revers-primer) bør ha nesten lik verdi, og helst identisk verdi.
Vi benyttet den gratis tilgjengelige primerdesign-programmvaren Primer-BLAST [12, 15] for å evaluere best-practice verdier for alle primere som er brukt i Corman-Drosten-rapporten (tabell 3). Vi forsøkte å finne en Tm-verdi på 60° C, samtidig som vi søkte etter den høyest mulige GC%-verdien for alle primere. En maksimal forskjell på 2° C i primerparene ble regnet som akseptabel. Da vi testet primerparene som er spesifisert i Corman-Drosten-rapporten (tabell 3), viste det seg en differanse på 10° C når det gjelder smeltetemperaturer Tm for primerpar 1 (RdRp_SARSr_F og RdRp_SARSr_R). Dette er enda en meget alvorlig feil som gjør protokollen ubrukelig som et spesifikt diagnostisk verktøy.
Ytterligere testing viste at bare primerparet som var designet for å amplifisere N-genet (N_Sarbeco_F og N_Sarbeco_R) oppnådde adekvat standard for å kunne brukes i en diagnostisk test, ettersom det har tilstrekkelig GC-innhold, og Tm-differansen mellom primere (N_Sarbeco_F and N_Sarbeco_R) er 1.85° C (mindre enn den avgjørende maksimale 2° C differansen). Det er viktig at dette er genet som verken var testet i virusprøvene (tabell 2), eller lagt vekt på som en bekreftende test. I tillegg til meget variable smeltetemperaturer og degenererte nukleotider i disse primer-sekvensene, er det en annen faktor som påvirker spesifisiteten til prosedyren: dNTPs (0.4 uM) er 2x høyere enn anbefalt for en høy-spesifikk amplifikasjon. Det er også tilsatt ytterligere magnesiumsulfat i reaksjonen. Denne fremgangsmåten, kombinert med en lav smeltetemperatur, kan føre til ikke-spesifikke amplifikasjoner. Når mer magnesium er påkrevet for qPCR bør spesifisiteten av assayet undersøkes nærmere.
Designfeilene som er beskrevet her er så alvorlige at det er svært lite sannsynlig at spesifikk amplifikasjon av genetisk materiale av SARS-CoV-2 vil skje når man benytter protokollen til Corman-Drosten-rapporten.
Tabell 3: GC-innhold av primere og prober (etter Corman-Drosten-rapporten; avvik fra optimalisert GC-innhold er uthevet). Panel 2 viser en tabell av alle best-practice-verdier vha. Primer-BLAST for alle primere og prober beskrevet i Corman-Drosten-rapporten, utført av Prof. Dr. Ulrike Kämmerer og hennes team.

3. Antall amplifikasjons-sykler
Det er bemerkelsesverdig at det ikke nevnes noe sted i Corman-Drosten-rapporten om en test er positiv eller negativ, eller i det hele tatt hvordan et positivt eller negativt resultat defineres. Disse typer virologiske diagnostiske tester må være basert på en SOP, inklusive et validert og fastsatt antall av PCR-sykluser (Ct-verdi/syklus-terskelverdi). Deretter blir en prøve vurdert som positiv eller negativ. Den maksimale rimelig pålitelige Ct-verdien er 30 sykluser. Over en Ct-verdi på 35 sykluser må det forventes et raskt økende antall falske positive.
PCR-data som er vurdert som positiv på grunnlag av en Ct-verdi på 35 sykluser er helt upålitelige.
Sitat fra Jafaar et al. 2020 [3]:
«Ved Ct = 35, altså den verdien som vi benyttet til å rapportere et positivt resultat for PCR, var <3% av kulturene positive.»
Med andre ord, det var ingen vellykket virus-påvisning av SARS-CoV-2 ved disse høye Ct-verdiene.
I tillegg viser andre vitenskapelige studier at bare ikke-smittsomme (døde) virus blir oppdaget ved Ct-verdier på 35 [22].
Mellom 30 og 35 er det en gråsone, der en positiv test ikke kan bestemmes med sikkerhet. Dette området bør ekskluderes. Man kunne naturligvis kjøre 45 sykler, som anbefalt i Corman-Drosten WHO-protokollen (figur 4), men da må man også definere en rimelig Ct-verdi (som ikke bør overstige 30). Men et analytisk resultat med en Ct-verdi på 45 er vitenskapelig og diagnostisk totalt meningsløst (en rimelig Ct-verdi bør ikke overstige 30). Alt dette bør formidles svært tydelig. Det er en vesentlig feil at Corman-Drosten-rapporten ikke nevner den maksimale Ct-verdien der en prøve utvetydig kan bli vurdert som et positivt eller negativt testresultat. Denne viktige syklus-terskelen er heller ikke til dags dato spesifisert i noen oppfølgende dokumenter (submissions).
Figur 4: RT-PCR-kit anbefaling i den offisielle Corman-Drosten WHO-protokollen [8]. Bare en «Cycler»-verdi (sykler) kan finnes, men uten korresponderende og vitenskapelig fornuftig Ct (cutoff-verdi). Verken denne eller noen annen syklusverdi kan finnes i selve Corman-Drosten-rapporten.

4. Biomolekylære valideringer
For å bestemme hvorvidt de amplifiserede produktene virkelig er SARS-CoV-2-gener, er biomolekylær validering av amplifiserte PCR-produkter av vesentlig betydning. For en diagnostiserende test er en slik validering et absolutt krav.
Validering av PCR-produkter bør gjennomføres enten ved å kjøre PCR-produktet i en 1% agarose-EtBr-gel sammen med en størrelses-indikator (DNA-linjal), slik at størrelsen av produktet kan estimeres. Størrelsen må da samsvare med den kalkulerte størrelsen av det amplifiserte produktet. Men det vil være enda bedre å sekvensere det amplifiserte produktet. Sistnevnte alternativ vil gi 100% sikkerhet om identiteten til det amplifiserte produktet. Det betyr at uten molekylær validering kan man ikke være sikker på identiteten til amplifiserte PCR-produkter. Med tanke på de alvorlige design-feilene som tidligere er beskrevet, kan derfor de amplifiserte PCR-produktene være hva som helst.
Noe som heller ikke er nevnt i Corman-Drosten-rapporten er saken med små fragmenter ved qPCR (omkring 100 bp): det kan enten være 1,5% agarosegel eller resultere fra akrylamidgel.
Det faktum at disse PCR-produktene ikke er blitt molekylær-teknisk validert er enda en slående feil ved protokollen. Det gjør at enhver test basert på den blir ubrukelig som et spesifikt diagnostisk verktøy for å identifisere SARS-CoV-2 viruset.
5. Positive og negative kontroller for å bekrefte/avkrefte spesifikk virusdeteksjon
Den ubekreftede antakelsen beskrevet i Corman-Drosten-rapporten er at SARS-CoV-2 er det eneste viruset fra den SARS-aktige betakoronavirus-gruppen som for tiden forårsaker infeksjoner i mennesker. Sekvensene som deres PCR-metode er basert på er in silico sekvenser, skaffet til veie av et laboratorium i Kina [23], fordi på det tidspunktet da PCR-testen ble utviklet av forfatterne, var det ikke tilgjengelig noe kontroll-materiale av smittsomt («levende») eller deaktivert SARS-CoV-2. PCR-testen ble derfor designet ved bruk av det kjente SARS-CoV som kontroll-materiale for Sarbeco-komponenten (Dr. Meijer, medforfatter av Corman-Drosten-rapporten i en epost-utveksling med Dr. Peter Borger) [2].
Alle individer som tester positivt med RT-PCR-testen, som beskrevet i Corman-Drosten-rapporten, er antatt å være positive for en infeksjon med SARS-CoV-2. Det er tre alvorlige feil i deres antakelse. For det første kan en positiv test for de RNA-molekylene som er beskrevet i Corman-Drosten-rapporten ikke likestilles med «virus-infeksjon». En positiv RT-PCR test indikerer bare forekomst av virale molekyler amplifisert vha. PCR. Som forklart under punkt 1d (over), var Corman-Drosten-protokollen ikke designet for å detektere et full-lengde virus, men bare et fragment av viruset. Vi har allerede konkludert med at dette klassifiserer testen som uegnet som en diagnostisk test for SARS-virus infeksjoner.
For det andre, og av stor betydning her, ble funksjonaliteten til den publiserte RT-PCR-testen ikke gjennomført og bekreftet ved hjelp av en positiv kontroll (isolert SARS-CoV-2 RNA), noe som er en essensiell vitenskapelig gullstandard.
For det tredje hevder Corman-Drosten-rapporten:
«For å vise at analysen kan detektere andre flaggermus-assosierte SARS-relaterte virus, brukte vi E-gen-analysen for å teste seks flaggermus-avledete ekskrement-prøver tilgjengelig fra Drexler et al. […] og Muth et al. […]. Disse virus-positive prøvene stammet fra europeiske rhinolophid flaggermus. Deteksjon av disse polygenetiske fremmede variantene innenfor den SARS-relaterte CoV-grenen antyder at alle asiatiske virus sannsynligvis vil bli detektert. Dette ville i teorien sikre bred sensitivitet og inkludere flere uavhengige virus-varianter fra et dyrereservoar.»
Dette utsagnet viser at E-genet som er brukt i RT-PCR testen, som beskrevet i Corman-Drosten-rapporten, ikke er spesifikt for SARS-CoV-2.
E-gen-primerne detekterer også et bredt spekter av andre SARS-virus. Koronavirusets genom er det største av alle RNA-virus som smitter mennesker, og de har alle sammen en svært lik molekylær struktur. Likevel har SARS-CoV og SARS-CoV-2 to meget spesifikke genetiske fingeravtrykk, som skiller dem fra andre koronavirus. For det første er en unik fingeravtrykk-sekvens (KTFPPTEPKKDKKKK) til stede i N-proteinet til SARS-CoV og SARS-CoV-2 [13,14,15]. For det andre inneholder verken SARS-CoV eller SARS-CoV-2 HE-proteinet, mens alle andre koronavirus har dette genet [13, 14]. Så for å spesifikt detektere SARS-CoV og SARS-CoV-2 PCR-produktet burde det nevnte området i N-genet vært valgt som amplifiserings-mål. En pålitelig diagnostisk test bør fokusere på dette spesifikke området i N-genet som en bekreftende test. PCR-assayet for dette N-genet ble ikke videre validert eller anbefalt som testgen i Corman-Drosten-rapporten, fordi det var «ikke så følsomt» med den originale SARS-CoV-proben [1].
I tillegg gjør fraværet av HE-genet i både SARS-CoV og SARS-CoV-2 dette genet til en ideell negativ kontroll for å ekskludere andre koronavirus. Corman-Drosten-rapporten inneholder ikke denne negative kontrollen, og heller ikke noen andre negative kontroller. PCR-testprotokollen i Corman-Drosten-rapporten inneholder derfor verken en unik positiv kontroll eller en negativ kontroll for å ekskludere forekomst av andre koronavirus. Dette er enda en alvorlig design-feil som klassifiserer testen som uegnet for diagnostisering.
6. Standard Operasjons-prosedyre (SOP) er ikke tilgjengelig
En standard operasjons-prosedyre (SOP) bør være tilgjengelig, som utvetydig spesifiserer de ovennevnte parameterene, slik at alle laboratorier er i stand til å sette opp identiske test-forhold. Det er vesentlig å ha en validert universell SOP, fordi det gjør det lettere å sammenlikne data internt og mellom forskjellige land. Det er meget viktig å utvetydig spesifisere alle primer-parametere. Dette ikke er blitt gjort .Videre er ikke Ct-verdien spesifisert for å indikere når en prøve bør regnes for å være positiv eller negativ. Det er heller ikke spesifisert når en prøve er vurdert å inneholde smittsomme SARS-CoV virus. Som vist tidligere kan testen ikke skille mellom virus og virus-fragmenter, derfor er Ct-verdien for å indikere positivitet avgjørende viktig. Denne Ct-verdien burde ha vært spesifisert i en SOP og vist on-line, slik at alle laboratorier som utfører denne testen benytter nøyaktig samme grenseverdier. Når en slik SOP ikke finnes, tyder det på mangelfull forskning. Laboratoriene står dermed fritt til å foreta testen slik de definerer hensiktsmessig, noe som resulterer i en enorm mengde variasjon. Laboratorier over hele Europa blir stående igjen med med en mengde spørsmål; hvilke primerne skal bestilles? Hvilke nukleotider for å fylle de udefinerte stedene? Hvilken Tm verdi skal velges? Hvor mange PCR-sykluser skal kjøres? Ved hvilken Ct-verdi er prøven positiv? Og når er den negativ? Og hvor mange gener skal testes? Skal alle gener testes, eller bare E- og RpRd-genene som vist i tabell 2 i Corman-Drosten-rapporten? Bør N-genet også testes? Og hva er deres negative kontroll? Hva er deres positive kontroll?
Slik protokollen er beskrevet er den dessverre svært vag og feilaktig i sitt design, slik at man kan gå i mange forskjellige retninger. Det er åpenbart at det ikke finnes noen standardisering eller en SOP, dermed er uklart hvordan denne testen skal implementeres.
7. Konsekvenser av feilene omtalt under 1-5: falsk positive resultater
RT-PCR testen som beskrevet i Corman-Drosten-rapporten inneholder så mange molekylære biologiske design-feil (se 1-5) at det ikke er mulig å oppnå udiskutable resultater. Det er uunngåelig at denne testen kommer til å generere et voldsomt stort antall såkalte «falske positive». Definisjonen på en falsk positiv er en negativ prøve, som først skårer positivt, men som skårer negativ etter re-testing med den samme testen. Falske positive er feilaktige positive testresultater, det vil si negative prøver som tester positivt. Og dette er faktisk hva man finner i Corman-Drosten-rapporten. På side 6 i PDF-manuskriptet viser forfatterne at selv under godt kontrollerte forhold oppstår en betydelig prosentdel falske positiver med denne testen:
«I fire individuelle testreaksjoner ble først svak reaktivitet observert, men de samme prøvene var negative ved re-testing med den samme analysen. Disse signalene var ikke assosiert med noe spesielt virus; for hvert virus der innledende positiv reaktivitet fant sted, var det andre prøver som inneholdt det samme viruset ved en høyere konsentrasjon, men som ikke testet positivt. Gitt resultatene fra den omfattende tekniske utprøvningen som er beskrevet ovenfor, ble det konkludert med at den innledende reaktiviteten ikke skyldtes kjemisk ustablilitet fra sanntid PCR-prober, men mest sannsynlig var grunnet i håndteringsproblemer forårsaket av den raske introduseringen av nye diagnostiske tester og kontroller i løpet av denne evalueringsstudien.» [1]
Den første setningen i dette utdraget er et klart bevis på at PCR-testen som beskrevet i Corman-Drosten-rapporten genererer falske positive resultater. Selv under godt kontrollerte forhold i Charité-sykehusets state-of-the-art laboratorium er 4 av 310 primær-tester per definisjon falske positive. Fire negative prøver testet først positivt, og ble deretter negative ved re-testing. Dette er det klassiske eksempel på en falsk positiv. I dette tilfellet identifiserer forfatterne dem ikke som falske positiver, noe som er intellektuelt uhederlig.
En annen avslørende observasjon i det ovenstående utdraget er at forfatterne bortforklarer de falske positive som »håndteringsproblemer forårsaket av den raske introduseringen av nye diagnostiske tester». Forestill deg laboratorier som nå er nødt til å innføre testen uten all den nødvendige informasjonen som normalt er omtalt i en SOP.
8. Corman-Drosten-rapporten var ikke fagfellevurdert
Før formell publisering i et akademisk tidsskrift blir vitenskapelige og medisinske artikler tradisjonelt sertifisert ved hjelp av «fagfellevurdering». I denne prosessen mottar tidsskriftets redaktører råd fra forskjellige eksperter («dommere») som har vurdert manuskriptet og som kan identifisere svakheter i dens antakelser, metoder og konklusjoner. Et tidsskrift vil normalt bare publisere en artikkel etter at redaktørene har forsikret seg om at forfatterne har tatt stilling til dommernes bemerkninger, og at data som presenteres støtter opp under rapportens konklusjoner. Denne prosessen er godt beskrevet når det gjelder Eurosurveillance [16].
Corman-Drosten-rapporten ble innsendt til Eurosurveillance 21. januar 2020 og ble godkjent for publisering 22. januar 2020. 23. januar 2020 var artikkelen on-line. 13. januar 2020 ble versjon 1.0 av protokollen publisert på WHOs offisielle nettsted [17], oppdatert 17. januar 2020 som dokumentversjon 2.1 [18], til og med før Corman-Drosten-rapporten ble publisert 23. januar hos Eurosurveillance.
Fagfellevurdering er vanligvis en tidkrevende prosess, ettersom minst to eksperter på fagområdet må foreta en kritisk gjennomgang og gi kommentarer til det innsendte vitenskapelig arbeide. Etter vår mening er det foreliggende arbeide ikke blitt fagfellevurdert. 24 timer er rett og slett ikke nok for å gjennomføre en grundig fagfellevurdering. Vår konklusjon støttes av det faktum at en stor mengde svært alvorlige design-feil ble oppdaget av oss, noe som gjør PCR-testen fullstendig uegnet til å være et diagnostisk verktøy for å identifisere SARS-CoV-2 viruset. Enhver molekylærbiolog som er kjent med RT-PCR design ville enkelt ha observert de grove feilene som finnes i Corman-Drosten-rapporten, helst før den egentlige vurderingsprosessen. 26. oktober 2020 ba vi Eurosurveillance om å sende oss en kopi av fagfellevurderingsrapporten. Til dags dato har vi ikke mottatt denne rapporten, og i et brev fra 18. november 2020 avslo ECDC – som eier Eurosurveillance – å gi oss tilgang, uten å oppgi tilstrekkelige vitenskapelige grunner for sin beslutning. Tvert imot skriver de at «offentliggjøring ville undergrave hensikten med vitenskapelige undersøkelser» [24].
9. Forfattere som redaktører
Et siste punkt gir alvorlig bekymring. Det viser seg at to av forfatterne av Corman-Drosten-rapporten, Christian Drosten og Chantal Reusken, også er medlemmer av redaksjonsrådet i dette tidsskriftet [19]. Det er derfor en alvorlig interessekonflikt som forsterker mistanken om at rapporten ikke ble fagfellevurdert. Det gir inntrykk av at den raske publiseringen var mulig rett og slett fordi forfatterne også var medlemmer av redaksjonsrådet i Eurosurveillance. Slik praksis blir ansett som kompromitterende for vitenskapelig integritet.
OPPSUMMERENDE OVERSIKT OVER FEIL SOM ER OPPDAGET I RAPPORTEN
Corman-Drosten-rapporten inneholder følgende spesifikke feil:
- Det foreligger ingen oppgitt grunn til å benytte disse ekstremt høye konsentrasjonene av primerne i protokollen. De omtalte konsentrasjonene fører til økt ikke-spesifikke bindinger og PCR-produktamplifikasjoner, noe som gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.’
- Seks uspesifiserte posisjoner kommer til å medføre en enorm variasjon i laboratorie-implementering av testen ute i den virkelige verden; den forvirrende uspesifiserte beskrivelsen i Corman-Drosten-rapporten er ikke egnet som SOP, noe som gjør at testen ikke er brukbar som et spesifikt diagnostisk verktøy for å identifisere SARS-CoV-2-viruset.
- Testen kan ikke skille mellom hele virus og virale fragmenter. Testen kan derfor ikke benyttes til diagnostisering av intakte (smittsomme) virus. Dette gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset og gir ikke grunnlag til å kunne trekke en konklusjon om en infeksjon foreligger.
- En differanse på 10° C når det gjelder smeltetemperaturen, Tm, for primerpar-1 (RdRp_SARSr_F og RdRp_SARSr_R) gjør også testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.
- En alvorlig feil er utelatelsen av å angi en Ct-verdi for å vurdere når en prøve er positiv eller negativ. Denne Ct-verdien lar seg heller ikke finne i innsendte oppfølgende dokumenter, noe som gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2-viruset.
- PCR-produktene er ikke blitt validert på molekylært nivå. Dette faktum gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.
- PCR-testprotokollen inneholder verken en unik positiv kontrollprøve for evaluering av testens spesifisitet for SARS-CoV-2 eller en negativ kontroll for å ekskludere andre koronavirus, noe som gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.
- Testens design i Corman-Drosten-rapporten er så vag og mangelfull at testen kan gjennomføres på tusen forskjellig vis; ingenting er standardisert og det foreligger ingen SOP. Dette stiller alvorlige spørsmålstegn ved testens vitenskapelige gyldighet og gjør den uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.
- Mest sannsynlig ble Corman-Drosten-rapporten ikke fagfellevurdert, noe som gjør testen uegnet som spesifikt diagnostisk verktøy for identifisering av SARS-CoV-2 viruset.
- Vi finner alvorlige interessekonflikter for minst fire forfattere, i tillegg til at to av forfatterne av Corman-Drosten-rapporten (Christian Drosten og Chantal Reusken) er medlemmer av redaksjonsrådet i Eurosurveillance. En ny interessekonflikt ble tilføyd 29. juli 2020 (Olfert Landt er CEO i TIB-Molbiol; Marco Kaiser er seniorforsker ved GenExpress og arbeider også som vitenskapelig rådgiver for TIB-Molbiol), noe som ikke ble opplyst i den opprinnelige utgaven (og som fortsatt mangler i PubMed versjonen); TIB-Molbiol er selskapet som var «først ute» med å produsere PCR-kits (Light Mix), basert på protokollen som ble publisert i Corman-Drosten manuskriptet, og som i følge deres egne ord distribuerte disse PCR test-settene allerede før publikasjonen overhodet var innsendt [20]; dessuten unnlot Victor Corman og Christian Drosten å nevne sin andre tilhørighet: det kommersielle test-laboratoriet “Labor Berlin”. Både Victor Corman og Christian Drosten er ansvarlige for virus-diagnostisering i den nevnte firmaen [21], og firmaets fagområde, bl.a., er nettopp sanntid PCR-testing.
Basert på vår granskning av test-protokollen for å identifisere SARS-CoV-2 som beskrevet i Corman-Drosten-rapporten har vi oppdaget bekymringsverdige feil og iboende feilslutninger som gjør at SARS-CoV-2 PCR-testen er ubrukelig.
KONKLUSJON
Avgjørelsen om hvilke testprotokoller som blir publisert og gjort tilgjengelig for et bredt publikum ligger fullt og helt hos Eurosurveillance. Et vedtak om å erkjenne de åpenbare feilene i Corman-Drosten-rapporten vil ha fordelen av å i stor grad redusere menneskelige kostnader og lidelser i fremtiden.
Er det ikke i Eurosurveillance sin beste interesse å trekke tilbake denne publikasjonen? Vår konklusjon er klar. I lys av alle de enorme feil og mangler ved PCR-testprotokollen som er beskrevet her, konkluderer vi med at man ikke har mye valg gitt kravene om vitenskapelig integritet og ansvarlighet.
REFERANSER
[1] Corman Victor M, Landt Olfert, Kaiser Marco, Molenkamp Richard, Meijer Adam, Chu Daniel KW, Bleicker Tobias, Brünink Sebastian, Schneider Julia, Schmidt Marie Luisa, Mulders Daphne GJC, Haagmans Bart L, van der Veer Bas, van den Brink Sharon, Wijsman Lisa, Goderski Gabriel, Romette Jean-Louis, Ellis Joanna, Zambon Maria, Peiris Malik, Goossens Herman, Reusken Chantal, Koopmans Marion PG, Drosten Christian. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 2020;25(3):pii=2000045. https://doi.org/10.2807/1560-7917.ES.2020.25.3.2000045
[2] Email communication between Dr. Peter Borger & Dr. Adam Meijer: Supplementary Material
[3] Jafaar et al., Correlation Between 3790 Quantitative Polymerase Chain Reaction–Positives Samples and Positive Cell Cultures, Including 1941 Severe Acute Respiratory Syndrome Coronavirus 2 Isolates. https://academic.oup.com/cid/advance-article/doi/10.1093/cid/ciaa1491/5912603
[4] BBC, January 21st 2020: https://www.bbc.com/news/world-asia-china-51185836;
Archive: https://archive.is/0qRmZ
[5] Google Analytics – COVID19-deaths worldwide: https://bit.ly/3fndemJ
Archive: https://archive.is/PpqEE
[6] Laboratory testing for COVID-19 Emergency Response Technical Centre, NIVD under
China CDC March 15th, 2020: http://www.chinacdc.cn/en/COVID19/202003/P020200323390321297894.pdf
[7] Real-Time PCR Handbook Life Technologies: https://www.thermofisher.com/content/dam/LifeTech/global/Forms/PDF/real-time-pcr-
handbook.pdf
Nolan T, Huggett J, Sanchez E.Good practice guide for the application of quantitative PCR (qPCR) First Edition 2013
[8] Trestan Pillonel et al, Letter to the editor: SARS-CoV-2 detection by real-time RT-PCR: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7268274/
[9] Kurkela, Satu, and David WG Brown. “Molecular-diagnostic techniques.” Medicine 38.10
(2009): 535-540.
[10] Wolfel et al., Virological assessment of hospitalized patients with COVID-2019
https://www.nature.com/articles/s41586-020-2196-x
[11] Thermofischer Primer Dimer Web Tool: https://www.thermofisher.com/us/en/home/brands/thermo-scientific/molecular-biology/molecular-biology-learning-center/molecular-biology-resource-library/thermo-scientific-web-tools/multiple-primer-analyzer.html
Supplementary Material
[12] Primer-BLAST, NCBI – National Center for Biotechnology Information: https://www.ncbi.nlm.nih.gov/tools/primer-blast/
[13] Marra MA, Steven JMJ, Caroline RA, Robert AH, Angela BW et al. (2003) Science. The
Genome sequence of the SARS-associated coronavirus. Science 300(5624): 1399-1404.
[14] Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete
genome: https://www.ncbi.nlm.nih.gov/nuccore/MN908947
[15] Borger P. A SARS-like Coronavirus was expected but nothing was done to be prepared. Am J Biomed Sci Res 2020. https://biomedgrid.com/pdf/AJBSR.MS.ID.001312.pdf
https://www.researchgate.net/publication/341120750_A_SARS-
like_Coronavirus_was_Expected_but_nothing_was_done_to_be_Prepared;
Archive: https://archive.is/i76Hu
[16] Eurosurveillance paper evaluation / review process: https://www.eurosurveillance.org/evaluation
[17] Official recommendation of the Corman-Drosten protocol & manuscript by the WHO,published on January 13th 2020 as version 1.0 of the document:
https://www.who.int/docs/default-source/coronaviruse/wuhan-virus-assay-
v1991527e5122341d99287a1b17c111902.pdf; archive: https://bit.ly/3m3jXVH
[18] Official WHO-recommendation for the Corman / Drosten RT-qPCR-protocol, which
directly derives from the Eurosurveillance-publication, document-version 2-1, published on
17th January 2020: https://www.who.int/docs/default-source/coronaviruse/protocol-v2-
1.pdf?sfvrsn=a9ef618c_2
[19] Eurosurveillance Editorial Board, 2020: https://www.eurosurveillance.org/upload/site-
assets/imgs/2020-09-Editorial%20Board%20PDF.pdf;
Archive: https://bit.ly/2TqXBjX
[20] Instructions For Use LightMix SarbecoV E-gene plus EAV Control, TIB-Molbiol & Roche
Molecular Solutions, January 11th 2020: https://www.roche-as.es/lm_pdf/MDx_40-0776_96_Sarbeco-E-
gene_V200204_09164154001 (1).pdf
Archive, timestamp – January 11th 2020: https://archive.is/Vulo5;
Archive: https://bit.ly/3fm9bXH
[21] Christian Drosten & Victor Corman, responsible for viral diagnostics at Labor Berlin:
https://www.laborberlin.com/fachbereiche/virologie/
Archive: https://archive.is/CDEUG
[22] Tom Jefferson, Elizabeth Spencer, Jon Brassey, Carl Heneghan Viral cultures for COVID-
19 infectivity assessment. Systematic review. Systematic review doi:
https://doi.org/10.1101/2020.08.04.20167932 https://www.medrxiv.org/content/10.1101/2020.08.04.20167932v4
[23] Kim et al.,The Architecture of SARS-CoV-2 Transcriptome:
https://www.sciencedirect.com/science/article/pii/S0092867420304062
[24] ECDC reply to Dr. Peter Borger, 18th November 2020:
Supplementary Material
[25] Prof. Dr. Ulrike Kämmerer & team, survey & Primer-BLAST table:
Supplementary Material
Ytterligere literatur:
Beskrivelse av RT-PCR RKI Germany, side 10 i denne lenken:
https://www.rki.de/DE/Content/Gesundheitsmonitoring/Gesundheitsberichterstattung/GBE
DownloadsJ/JoHM_S5_2020_Studienprotokoll_CORONA_MONITORING_lokal.pdf?__blob=p
ublicationFile
Forfatternes tilhørighet:
1) Dr. Pieter Borger (MSc, PhD), Molecular Genetics, W+W Research Associate, Lörrach, Germany
2) Rajesh Kumar Malhotra (Artist Alias: Bobby Rajesh Malhotra), Former 3D Artist / Scientific Visualizations at CeMM – Center for Molecular Medicine of the Austrian Academy of Sciences (2019-2020), University for Applied Arts – Department for Digital Arts Vienna, Austria
3) Dr. Michael Yeadon BSs(Hons) Biochem Tox U Surrey, PhD Pharmacology U Surrey. Managing Director, Yeadon Consulting Ltd, former Pfizer Chief Scientist, United Kingdom
4) Dr. Clare Craig MA, (Cantab) BM, BCh (Oxon), FRCPath, United Kingdom
5) Kevin McKernan, BS Emory University, Chief Scientific Officer, founder Medical Genomics, engineered the sequencing pipeline at WIBR/MIT for the Human Genome Project, Invented and developed the SOLiD sequencer, awarded patents related to PCR, DNA Isolation and Sequencing, USA
6) Prof. Dr. Klaus Steger, Department of Urology, Pediatric Urology and Andrology, Molecular Andrology, Biomedical Research Center of the Justus Liebig University, Giessen, Germany
7) Dr. Paul McSheehy (BSc, PhD), Biochemist & Industry Pharmacologist, Loerrach, Germany
8) Dr. Lidiya Angelova, MSc in Biology, PhD in Microbiology, Former researcher at the National Institute of Allergy and Infectious Diseases (NIAID), Maryland, USA
9) Dr. Fabio Franchi, Former Dirigente Medico (M.D) in an Infectious Disease Ward, specialized in “Infectious Diseases” and “Hygiene and Preventive Medicine”, Società Scientifica per il Principio di Precauzione (SSPP), Italy
10) Dr. med. Thomas Binder, Internist and Cardiologist (FMH), Switzerland
11) Prof. Dr. med. Henrik Ullrich, specialist Diagnostic Radiology, Chief Medical Doctor at the Center for Radiology of Collm Oschatz-Hospital, Germany
12) Prof. Dr. Makoto Ohashi, Professor emeritus, PhD in Microbiology and Immunology, Tokushima University, Japan
13) Dr. Stefano Scoglio, B.Sc. Ph.D., Microbiologist, Nutritionist, Italy
14) Dr. Marjolein Doesburg-van Kleffens (MSc, PhD), specialist in Laboratory Medicine (clinical chemistry), Maasziekenhuis Pantein, Beugen, The Netherlands
15) Dr. Dorothea Gilbert (MSc, PhD), PhD Environmental Chemistry and Toxicology. DGI Consulting Services, Oslo, Norway
16) Dr. Rainer J. Klement, PhD. Department of Radiation Oncology, Leopoldina Hospital Schweinfurt, Germany
17) Dr. Ruth Schruefer, PhD, human genetics/ immunology, Munich, Germany,
18) Dra. Berber W. Pieksma, General Practitioner, The Netherlands
19) Drs. Jan Bonte (GJ), Consultant Neurologist, The Netherlands
20) Dr. Bruno H. Dalle Carbonare (Molecular biologist), IP specialist, BDC Basel, Switzerland
21) Dr. Kevin P. Corbett, MSc Nursing (Kings College London) PhD (London South Bank) Social Sciences (Science & Technology Studies) London, England, United Kingdom
22) Prof. Dr. Ulrike Kämmerer, specialist in Virology / Immunology / Human Biology / Cell Biology, University Hospital Würzburg, Germany
Forfatternes bidrag:
PB: Planlegging og gjennomføring av analyser og forskning, konseptualisering av manuskriptet.
BRM: Planlegging og gjennomføring av analyser og forskning, konseptualisering av figurer og manuskriptet.
MY: Korrekturlesning av analyser og forskning.
KMcK: Gjennomføring av analyser og forskning, konseptualisering av manuskriptet.
KS: Gjennomføring av analyser og forskning.
PMcS: Korrekturlesning av analyser og forskning.
LA: Korrekturlesning av analyser og forskning.
FF: Korrekturlesning av analyser og forskning.
TB: Korrekturlesning av analyser og forskning.
HU: Korrekturlesning av analyser og forskning.
MO: Korrekturlesning av analyser og forskning.
SS: Korrekturlesning av analyser og forskning.
MDvK: Korrekturlesning av analyser og forskning.
DG: Korrekturlesning av analyser og forskning.
RJK: Korrekturlesning av analyser og forskning.
RS: Korrekturlesning av analyser og forskning, samt manuskriptet.
BWK: Korrekturlesning av analyser og forskning.
RvV: Korrekturlesning av analyser og forskning.
JB: Korrekturlesning av analyser og forskning.
KC: Korrekturlesning av analyser og forskning.
UK: Planlegging og gjennomføring av analyser og forskning, konseptualisering av manuskriptet.
Ytterligere korrekturlesere:
Saji N Hameed, Environmental Informatics, University of Aizu, Tsuruga, Ikki-machi, Aizuwakamatsu-shi, Fukushima, Japan
Howard R. Steen, MA Chem. Eng. Cantab, Former Research Manager, Germany
Tillegg
Oppdatert 2.12.2020:
Forfatterbidrag Dr. Michael Yeadon endret til:
Korrekturlesning av analyser og forskning.
Forfatters tilknytning Kevin Mckernan endret til:
Medicinal Genomics.
Oppdatering 5.3.2021
Co-signering tittel endret til:
Drs. Jan Bonte
En stavefeil er nå rettet.
Den feliaktige bruk av PCR-test (og «forsterkning»/sykluser)
https://www.faktisk.no/artikler/jy235/10-sporsmal-og-svar-om-pcr-testene